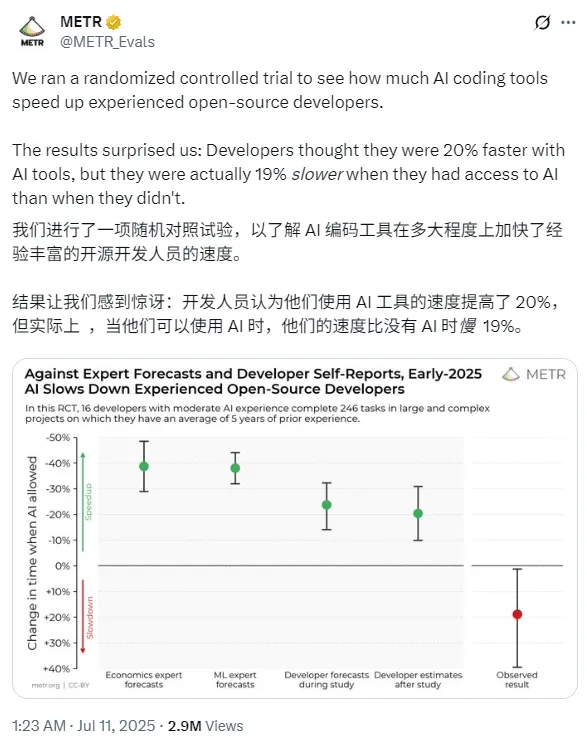
? 产品介绍
宾夕法尼亚大学合成生物工程实验室(César de la Fuente团队)专注AI驱动的自然分子挖掘,从灭绝生物DNA、动物毒液到古菌,突破传统抗生素研发瓶颈。其开发的APEX深度学习平台已累计发现386种毒液抗菌肽(91.4%验证有效)及上万种古菌抗菌化合物,成为新型抗生素研发的“加速引擎” 。
? 适用人群
- 医药研发企业:需突破现有抗生素结构的创新管线
- 微生物学家:极端环境微生物活性成分研究者
- AI药物发现团队:需提升候选分子筛选效率的实验室
- 公共卫生机构:应对抗生素耐药性危机的决策部门
⚙️ 核心功能
| 功能 | 技术实现原理说明 | 应用价值 |
|---|---|---|
| 多源数据整合 | 整合古菌基因组库+致病菌耐药性数据库 | 构建靶向性抗菌分子筛选网络 |
| 抗菌活性预测 | 基于序列-功能关系的深度神经网络(APEX 1.1) | 预测古菌肽抑制细菌生长的概率 |
| 跨膜作用模拟 | 电荷分布与膜穿透性耦合建模 | 识别可穿透细菌胞壁的分子 |
| 低毒性筛选 | 人体细胞毒性多任务学习模型 | 排除90%高副作用候选物 |
| 结构新颖性评估 | 对抗生成网络(GAN)对比已知抗菌肽库 | 确保发现全新化学结构 |
? 技术原理
团队采用 “三级漏斗式”筛选架构:
- 初级扫描:
升级版APEX模型对233种古菌的蛋白质组进行扫描,通过预训练识别抗菌肽的电荷分布模式(+2至+7净电荷)和疏水性特征,初筛12,000+候选肽 。 - 机制预判:
利用电生理活性模型模拟化合物与细菌内膜的相互作用,锁定通过“内部断电机制”干扰细菌电信号的分子(传统抗生素仅5%具此特性)。 - 活体验证:
对80种高评分古菌素进行耐药菌抑制实验,93%有效;其中3种进入小鼠试验,1种(古菌素-B7)对院内耐药菌的抑制率与多黏菌素B相当,且无肾毒性反应 。
? 突破点:古菌素通过扰乱细菌质子梯度阻断能量供应,而非常规的细胞膜破坏机制,大幅降低耐药性演化速度 。
?️ 工具使用技巧
- 优先扫描极端环境古菌:
深海热泉、盐碱湖的古菌(如Haloferax volcanii)含更多高电荷抗菌肽,APEX预测准确率提升19% 。 - 参数调优建议:
将模型阈值设为MIC≤32 μmol/L(中位数),可排除95%无效肽段 。 - 跨物种验证策略:
对APEX筛选出的古菌素进行哺乳动物细胞膜穿透测试,提前规避溶血风险 。
? 访问地址
? APEX开源代码与古菌素数据库:
GitHub项目:github.com/PennBioAI/APEX_Archaea
(含预训练模型、古菌蛋白质组数据集及合成指南)
? 站长锐评:当人类在实验室苦寻新抗生素时,AI正从沸腾的酸液和深海热泉中搬救兵——这些存活了30亿年的古菌,或许就是破解耐药困局的“终极钥匙”。?
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章

暂无评论...