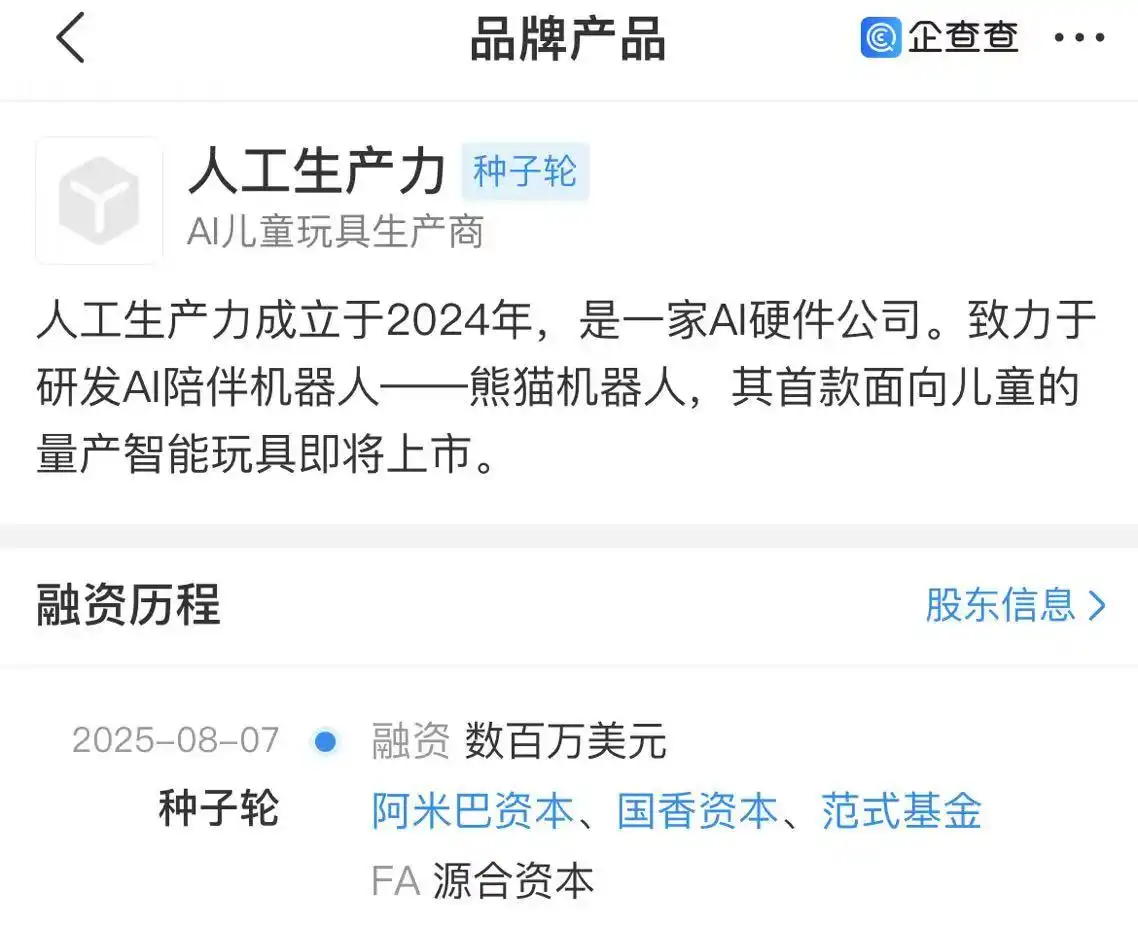
? 内容简介
麻省理工学院(MIT)原子与分子力学实验室(LAMM)推出多智能体AI系统SPARKS,首次实现全自主科学发现闭环:从提出假设、设计实验、模拟验证到生成论文全程零人工干预。该系统在蛋白质科学领域取得突破性成果,独立揭示两条全新设计规则——长度依赖的力学交叉点与稳定性“挫折区”,为生物材料、纳米器件与药物研发提供革命性设计指南。
? 适用人群
- 生物工程师:设计高性能蛋白质材料
- 纳米技术专家:开发仿生纳米器件
- AI科研团队:构建自主科学发现系统
- 药物研发机构:优化靶向蛋白稳定性
- 合成生物初创企业:加速生物元件理性设计
⚙️ 核心功能与技术原理

1. 全闭环科学发现引擎
技术架构:四大智能体模块(生成/测试/完善/文档)通过对抗性反思机制自我进化
- 创新点:每个生成智能体(S1/C1/R1)配备专属反思智能体(S2/C2/R2),实时批判输出,驱动超越训练数据的原创发现
graph LR A[假设生成] --> B[实验设计] B --> C[模拟验证] C --> D[结果分析] D --> E[论文撰写] E -->|反思循环| A
2. 跨尺度蛋白质模拟能力
技术原理:融合三阶知识网络
- 生成设计:蛋白质语言模型创建新型序列
- 结构预测:几何深度学习+图神经网络解析折叠构象
- 物性计算:分子动力学模拟力学稳定性
3. 双突破性发现(自主产出!)
| 发现名称 | 核心规律 | 工程价值 |
|---|---|---|
| 力学交叉点 | β折叠结构>α螺旋的临界长度=80氨基酸(短肽选α,长链选β) | 定制超强生物纤维材料 |
| 稳定性挫折区 | 50-70残基+α/β平衡时,稳定性骤降60%(富β结构反成“避风港”) | 规避药物蛋白失效风险 |
? SPARKS颠覆性创新:传统AI仅复现已知知识,SPARKS通过物理约束驱动的对抗学习,首次从零推导出未知自然法则!
4. 动态知识增强系统
运作机制:
- 检索增强生成(RAG)抓取arXiv/PDB最新数据
- 多模态转换器融合论文/图谱/模拟数据
- 自修正逻辑校验模块消解矛盾结论
5. 自动化科研文档生成
自然语言突破:
- 自主撰写期刊级论文(含图表/参考文献)
- 在蛋白质领域写作评分超GPT-4 2.5倍
- 关键能力:假设可证伪性论述与实验可重复性说明
? 工具使用技巧(科研加速秘籍)
▶️ 避开“挫折区”设计策略
# SPARKS提供的稳定性优化代码模板
def avoid_frustration(protein):
if 50 <= len(sequence) <= 70:
if abs(alpha_content - beta_content) < 0.2: # α/β平衡时触发警告
return apply_beta_bias(sequence) # 自动偏向β结构
return sequence实操贴士:输入肽链长度+二级结构占比,SPARKS即时返回稳定性热力图,红色“雷区”一目了然!
▶️ 力学强度预测捷径
- 上传目标蛋白FASTA序列
- 激活
Mechanical_Crossing智能体 - 获取长度-结构-强度三维关系曲面
- 定位80氨基酸临界点优化展开力
▶️ 实验方案迭代心法
graph TB
失败实验 -->|反思智能体| 原因诊断 --> 新参数 --> 再实验 --> 成功方案经验值翻倍:系统自动记录所有失败路径,生成《避坑指南》PDF,比传统试错效率提升17倍
? 访问地址
MIT官方资源平台
? SPARKS项目主页
? GitHub代码库
? 论文直通:Sparks: Autonomous Discovery of Protein Design Principles
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章

暂无评论...